iBOL2017
Disse resultatene, som ble presentert som poster 422 på den syvende IBOL-konferansen, gir et øyeblikksbilde på hvor langt vi har kommet i å bygge et kvalitetssikret strekkodebibliotek for marine virvelløse dyr (børstemark, pigghuder, krepsdyr, nesledyr, bløtdyr etc.) i fra Norge og tilstøtende områder (særlig Sverige, vi har samarbeidet mye med kollegaer i Gøteborg).
Før vi kunne sette i gang for alvor, måtte vi skaffe egnet materiale. Marint museumsmateriale er stort sett fiksert på formalin, som ødelegger DNA. Vi har derfor arrangert og deltatt på mange innsamlingstokt, og samarbeider også med store prosjekter som MAREANO og ulike miljøkartleggingsaktører for å skaffe prøver. Slik har vi de siste årene bygget opp store samlinger av spritfiksert materiale ved UM. Vi har også noen utfordringer med strekkodingsmetodikken; de såkalt «universelle» primerne som brukes i sekvenseringen har varierende suksessrate, og på enkelte dyregrupper vil det være nødvendig å inkludere andre markører (DNA-segmenter) enn standarden, som er cox1-segmentet.
Nå har vi – sammen med våre samarbeidspartnere – sendt inn over 8000 prøver til sekvensering. Av disse har vi fått sekvenser på 62 % av prøvene, og omkring 75 % av det vi kan kalle morfo-arter: dyr som vi enten kan identifisere til kjente arter basert på litteraturen, eller som vi midlertidig grupperer til «art A», «art B» etc. fordi vi ser at de skiller seg i fra de kjente artene.
Vi gjorde opp status for enkelte grupper i forhold til antallet kjente arter som finnes i Norge (basert på Artsdatabanken sine lister), og som du kan se er vi godt i gang på en del grupper – men det er fortsatt mye å ta tak i.
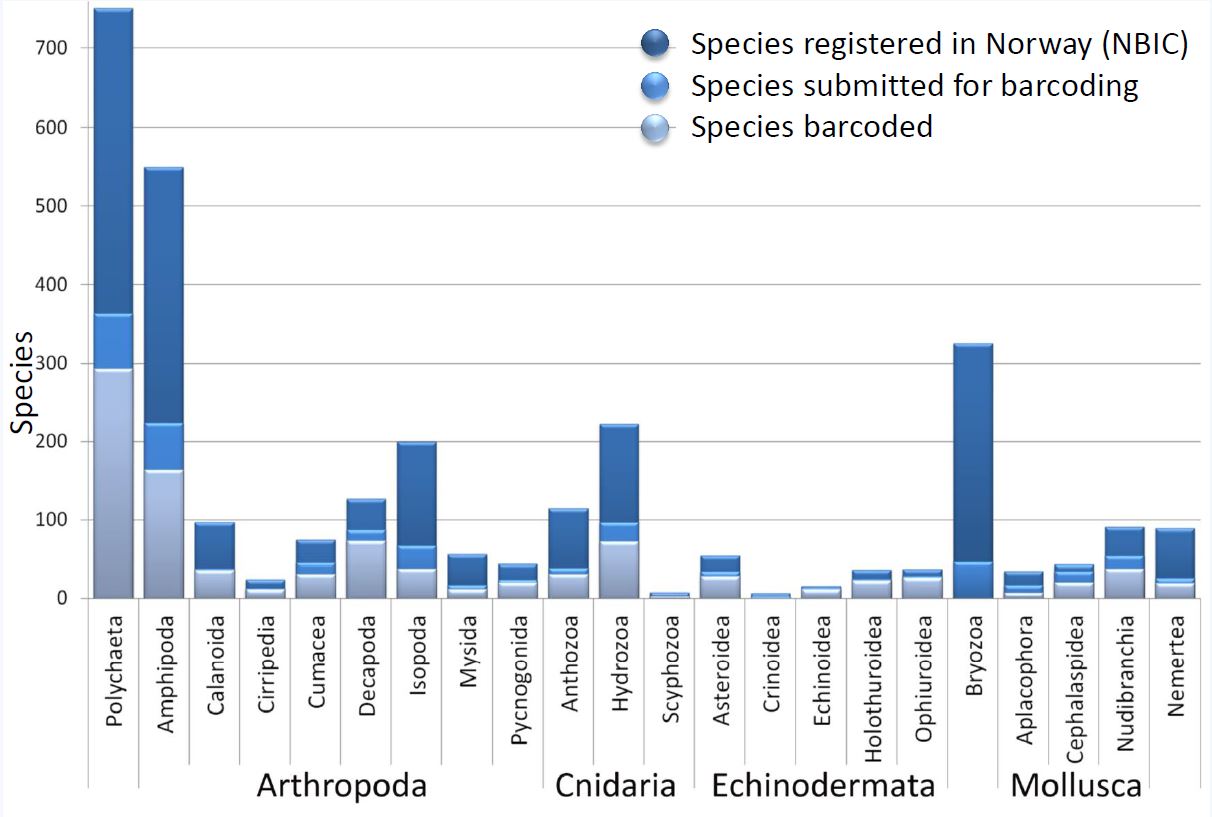
Det som ikke vises på denne figuren er de 2800 prøvene som vi ikke har fått strekkoder på med de gjeldene metodene, og de av prøvene som har fått strekkode, men som ikke har navn som korresponderer med Artsdatabanken sine lister over arter i Norge (dette kan skyldes f.eks. at arten ikke er registrert her, at den er ny for vitenskapen, eller at prøven vår ikke har blitt identifisert helt til art enda).
Her er to eksempler på observasjoner som bør følges opp med taksonomisk utredning:
Først ut er en stor sjøstjerne i fra slekten Diplopteraster. I fra Norge har tre individer av D. multipes blitt strekkodet. Når strekkodene for disse dyrene sammenlignes med andre i den store BOLD-basen så er de identiske med strekkoder som er produsert av andre marinbiologiske laboratorier, men som er identifisert med fem ulike artsnavn basert på morfologien.

Dette er verdt å se nærmere på – er de fem artene egentlig bare én vidt utbredt art, som vitenskapelige kulturer i ulike land har forstått som ulike arter, særegne for sin geografiske region? Eller er det den genetiske markøren vi har brukt som ikke klarer å skille mellom faktiske arter i denne gruppen? Slike spørsmål krever nærmere undersøkelser med taksonomiske studier, der det fysiske materialet som artsforståelsen er basert på kan studeres med kritisk blikk på den vitenskapelige litteraturen. I tillegg bør slike vurderinger av artsstatus helst ha data fra andre genetiske markører slik at både strekkoder og morfologiske karakterer kan vurderes i nytt lys. Noen ganger kan uoverensstemmende artsnavn for identiske strekkoder ganske enkelt skyldes menneskelig feil, – at den som navnsatte en strekkode tok feil i sin artsbestemmelse basert på morfologiske karakterer.
I det neste eksemplet har vi en antatt art med overraskende stor genetisk variasjon i det strekkodede materialet. Spørsmålet blir da om børstemarken Ophelina cylindricaudata i realiteten består av flere ulike arter. Det ser ut for at de genetiske gruppene som framkommer av strekkodedata kan kobles til ulike habitater som markene lever i.
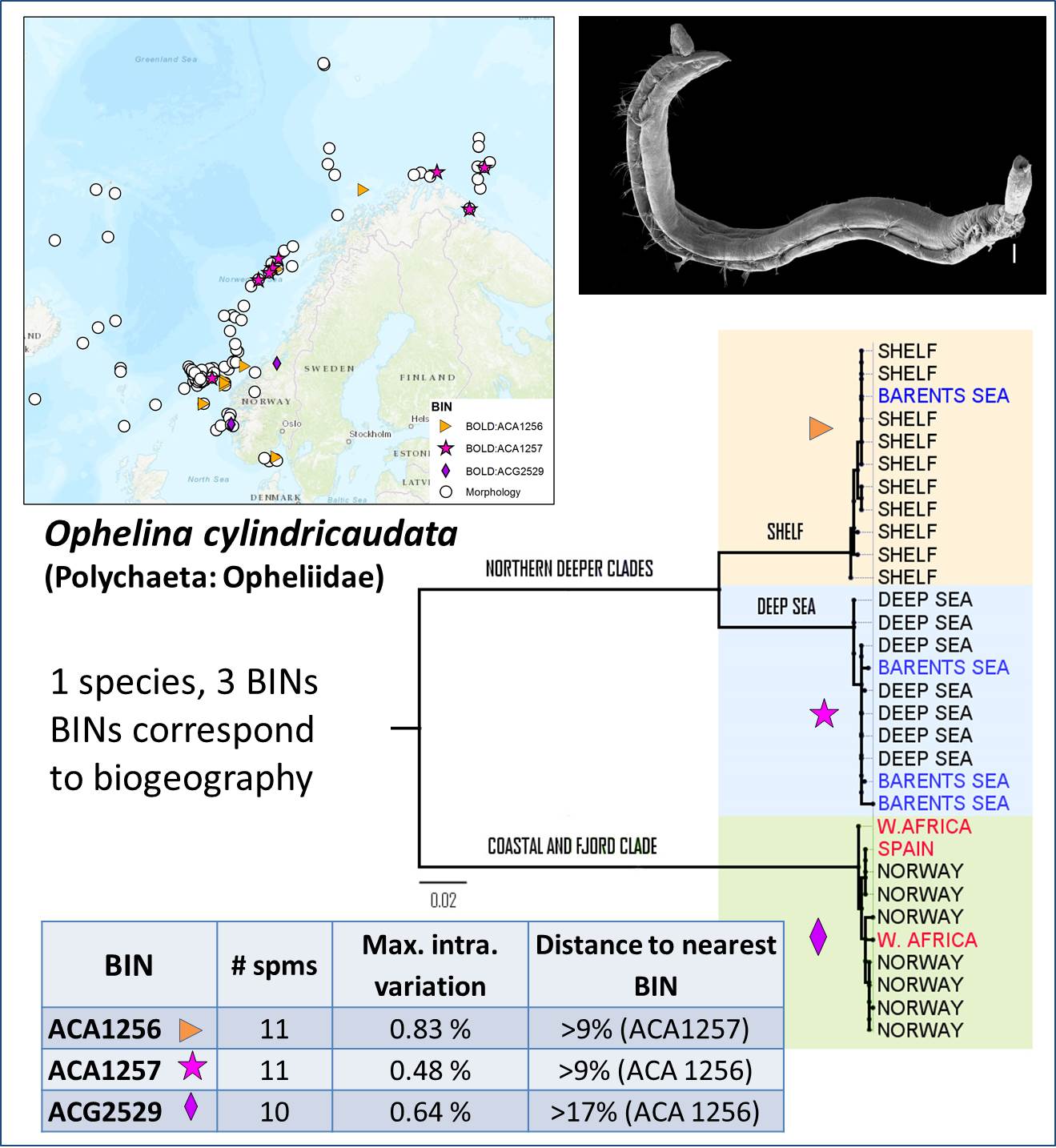
Igjen viser det seg et behov for nærmere taksonomiske, økologiske og evolusjonsbiologiske studier. Etterhvert er det ikke lengre uvanlig at nærmere studier av såkalt kryptiske arter først «avsløres» med strekkoder og ved nærmere studier kan vise seg å også ha morfologiske kjennetegn som skiller dem fra sine nærmeste slektninger.
Tilsvarende problemer som disse to eksemplene finnes det mange av. De viser at det er langt igjen til en presis og korrekt forståelse av artsmangfoldet i marine miljøer. Men med strekkoding kan vi finne hvor problemene befinner seg og slik sett identifisere behovene for videre arbeid. Selvfølgelig må vi heller ikke glemme at kvalitetssikrede strekkoder også gir vidtrekkende muligheter for så vel biologer som andre profesjoner for å identifisere organismetyper med stor presisjon, enten de foreligger som levende, døde, eller som DNA-spor.
Katrine Kongshavn, Jon A. Kongsrud, Tom Alvestad, Endre Willassen, Universitetsmuseet i Bergen, avdeling for naturhistorie.
Disse resultatene ble presentert på 7th International Barcode of Life Conference, Sør-Afrika, 2017.
Alfa-taksonomi er et forskningsområde som tar for seg den detaljerte kunnskapen om arter og artsgrupper, og inkluderer beskrivelse av nye arter, deres utbredelse og biologi. Med utgangspunkt i DNA-strekkodebiblioteket studerer vi en slekt av soppmygg (Mycetophilidae) som heter Allodia. I tillegg til DNA-strekkodene, ser vi også på andre genetiske markører og utseendet til Allodia-artene. Til sammen skal dette resultere i en revisjon, en slags opprydding, av hvilke arter som finnes i denne slekten og hvordan de er i slekt med hverandre og andre grupper av soppmygg.
Soppmyggene er en stor familie av tovinger (fluer, mygg og deres slektninger) som er spesielt artsrik i de nordlige delene av Europa og Amerika. Dette er trolig fordi larvene til de små tovingene lever av sopp, som også er svært artsrike og tallrike i disse områdene. Flere av artene i denne gruppen antas å ha en såkalt sirkumpolar utbredelse, altså en utbredelse tilknyttet det boreale skogbeltet og tundraen.
 Figur 1: Allodia sp. Foto: Karsten Sund, Naturhistorisk Museum ©.
Figur 1: Allodia sp. Foto: Karsten Sund, Naturhistorisk Museum ©.
I Norge og resten av Skandinavia har derfor disse små myggene fått mye oppmerksomhet fra forskere, og vi har en god oversikt over den Norske faunaen, blant annet gjennom Artsprosjektet. Den akkumulerte kunnskapen om norsk og skandinavisk soppmyggfauna har resultert i mange DNA-strekkoder fra identifiserte arter. Parallelt med at referansebiblioteket for DNA-strekkoder for soppmygg ble utviklet i Norge, hadde forskere i Canada, ved universitetet i Guelph en annen tilnærming til strekkoding av insekter. I stedet for å fokusere på DNA-strekkoder for identifiserte arter, som vi gjør, strekkoder de også en mengde uidentifiserte insekter fra Malaisefeller (en insektfelle som ligner et telt, samler mange forskjellige flyvende insekter), spesielt i Canada. Dette har resultert i en mengde DNA-strekkoder uten tilknytning til artsnavn.
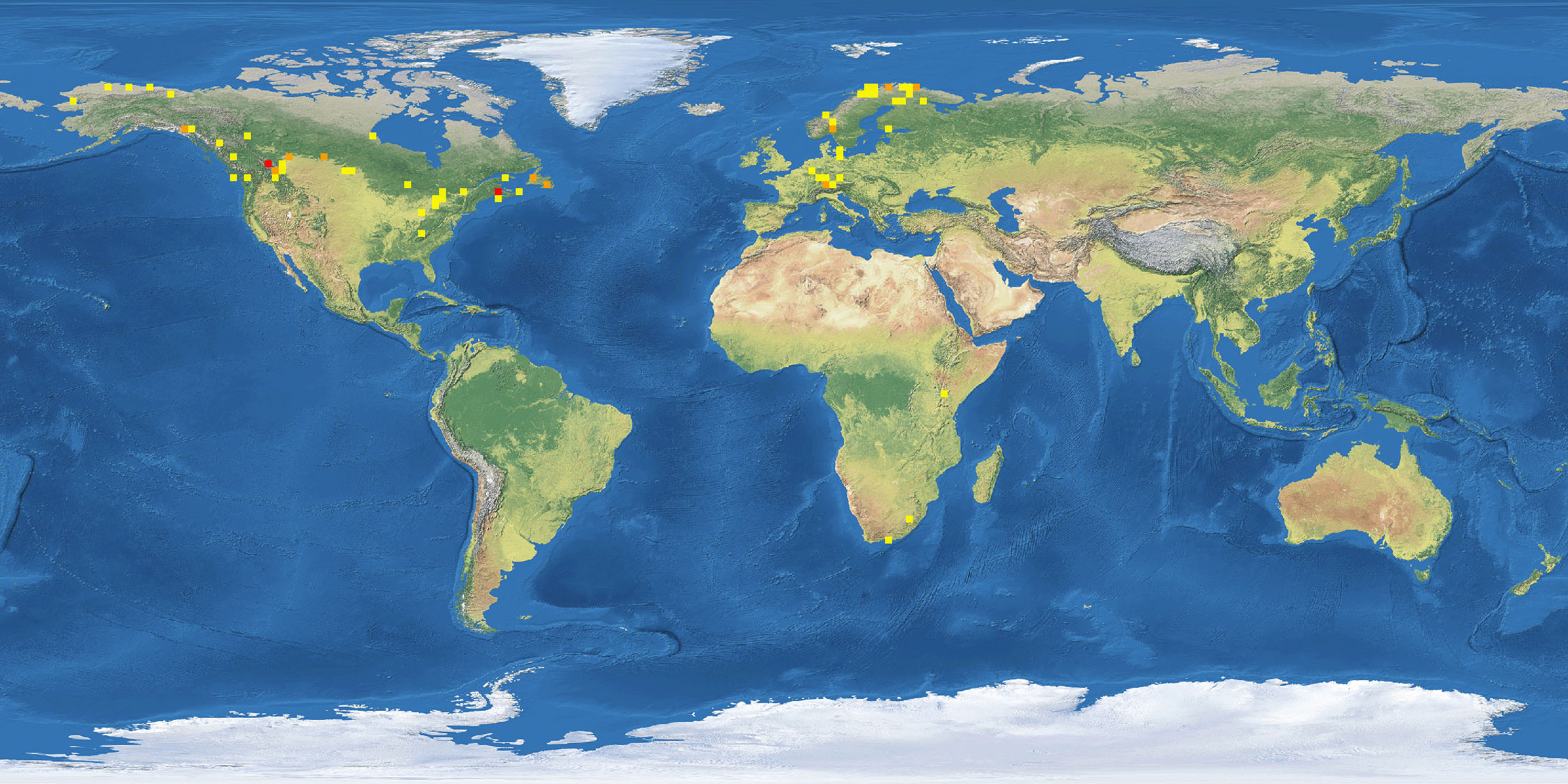 Figur 2: Kart fra BOLD som viser oversikten over hvor vi har DNA-strekkoder fra arter i slekten Allodia.
Figur 2: Kart fra BOLD som viser oversikten over hvor vi har DNA-strekkoder fra arter i slekten Allodia.
Samlet sett er dette et supert utgangspunkt for å studere ulike grupper av soppmygg mer detaljert, som for eksempel mangfold, utbredelsesmønstre og slektskap mellom arter. Vi kan da sammenligne både de identifiserte og ukjente DNA-strekkodene på tvers av landegrenser og regioner – perfekt for en gruppe som soppmyggene med nordlig utbredelse!
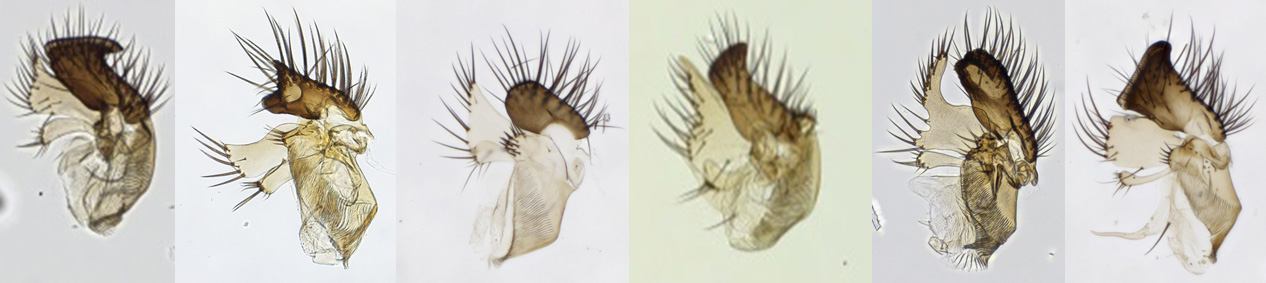 Figur 3: For å sammenligne utseende til ulike arter av soppmygg, må man se på genitaliene. Her er bilder av genitaliene til seks ulike arter Allodia. Foto: Trude Magnussen ©.
Figur 3: For å sammenligne utseende til ulike arter av soppmygg, må man se på genitaliene. Her er bilder av genitaliene til seks ulike arter Allodia. Foto: Trude Magnussen ©.
De foreløpige resultatene viser at flere arter finnes både i Canada og i Norge. I tillegg viser det seg at mange arter fra Canada trolig er nye for vitenskapen. Grunnen til at vi drar denne konklusjonen er at de dyrene som har ulik DNA-strekkode, også er ulike i genetiske markører fra kjerne DNA og i utseende. Interessant er det også at vi finner nye arter for vitenskapen i Norge! I det videre studiet skal vi se nærmere på slektskapet mellom artsgrupper i Norden og Canada.
Trude Magnussen, Stipendiat ved Naturhistorisk museum, Universitetet i Oslo
Disse resultatene ble presentert på 7th International Barcode of Life Conference, Sør-Afrika, 2017.
Artsprosjektet og NorBOL har hatt et godt samarbeid helt siden Artsprosjektet ble etablert i 2009.
Tilgang på materiale fra mindre kjente artsgrupper kan ofte være en begrensende faktor i arbeidet med å bygge opp et nasjonalt referansebibliotek med DNA-strekkoder. Gjennom den omfattende kartleggingen av arter som skjer i regi av Artsprosjektet, blir nytt materiale fra dårlig kjente arter identifisert av taksonomisk ekspertise og gjort tilgjengelig for DNA-sekvensering og registrering i BOLD. NorBOL gir prosjektledere veiledning i bruk av metoden og koordinerer arbeidet med DNA-strekkoding av arter samlet inn gjennom Artsprosjektet. Så langt har rundt halvparten av artene fra Norge som har fått en strekkode, opphav fra kartleggingen i Artsprosjektet.
 Noen av artene som har blitt registrert i Artsprosjektet. De fleste er også strekkodet gjennom NorBOL.
Noen av artene som har blitt registrert i Artsprosjektet. De fleste er også strekkodet gjennom NorBOL.
Tilgang på korrekt artsinformasjon er svært viktig for forskning og forvaltning. Ettersom DNA-strekkoding er et nyttig verktøy for å skille arter og oppdage ukjent diversitet, har samarbeidet mellom NorBOL og Artsprosjektet bidratt til både bedre kvalitet, flere datapunkter og større tilgjengelighet på artsrelatert informasjon i digitale tjenester. Begge prosjektene har hatt stor nytte av hverandre og sammen levert mer enn de kunne klart hver for seg.
Ingrid Salvesen, Artsdatabanken & Torbjørn Ekrem, NTNU Vitenskapsmuseet
Disse resultatene ble presentert på 7th International Barcode of Life Conference, Sør-Afrika, 2017.
Naturhistorisk museum i Oslo har ansvar for koordinering av strekkoding av norske lav, stilksporesopp og terrestriske insekter. Fra disse gruppene plukket vi ut tre eksempler på uoverensstemmelse mellom morfologi og strekkoder, som er oppdaget her ved museet, eller av våre samarbeidspartnere.

I lavslekten Calvitimela ble flere kryptiske arter oppdaget gjennom strekkoding, her vises C. melaleuca; hvorav den ene kryptiske arten viste seg å være søsterart til C. armeniaca. Det er ikke kjent hvilken art som er C. melaleuca s.str.
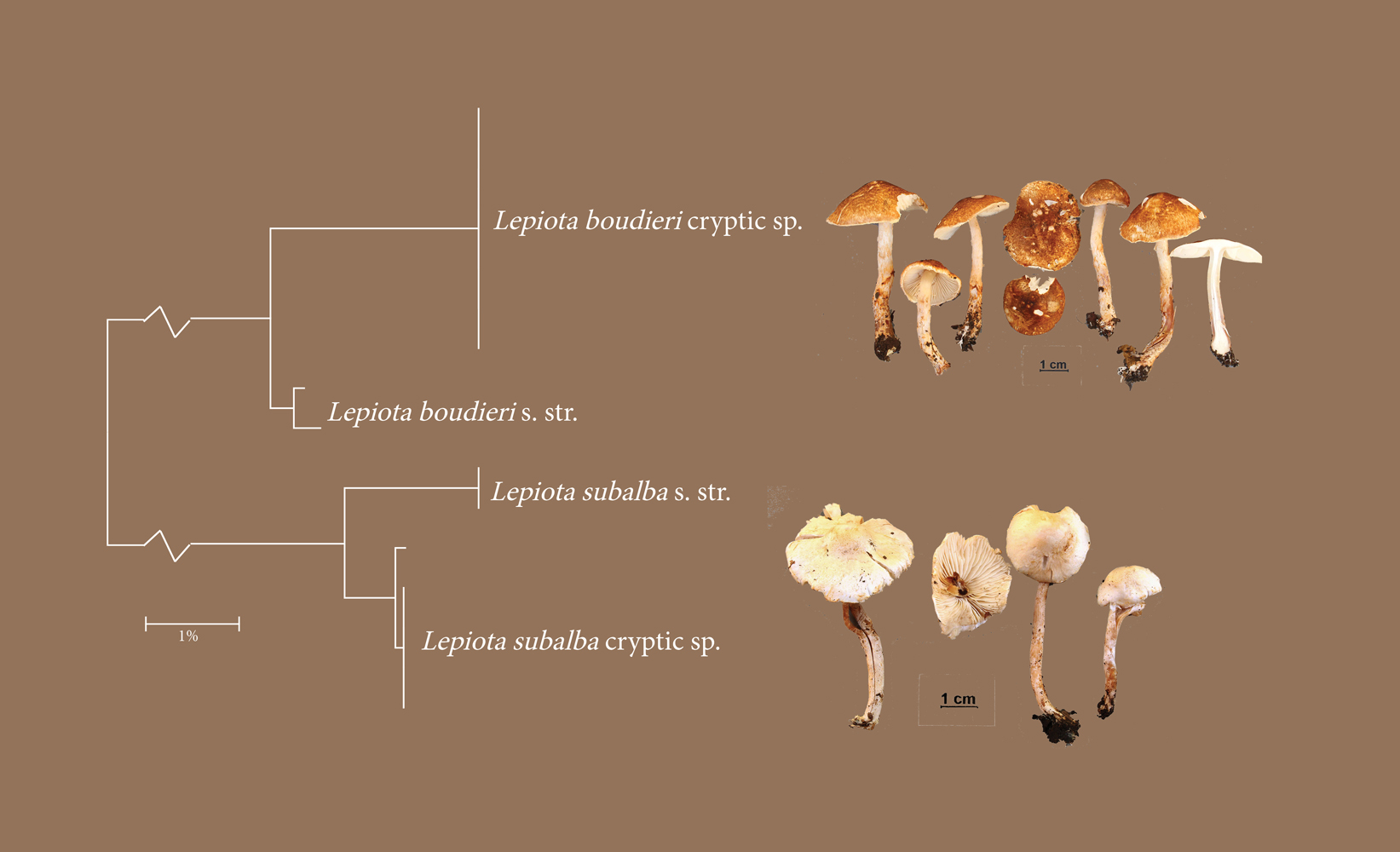
I soppslekten Lepiota (Agaricales) har de fleste morfologiske arter vist seg å bestå av to kryptiske arter. Her eksemplifisert med L. boudieri og L. subalba. Med denne kunnskapen vil sannsynligvis nærmere studier avsløre morfologiske kjennetegn som skiller dem.
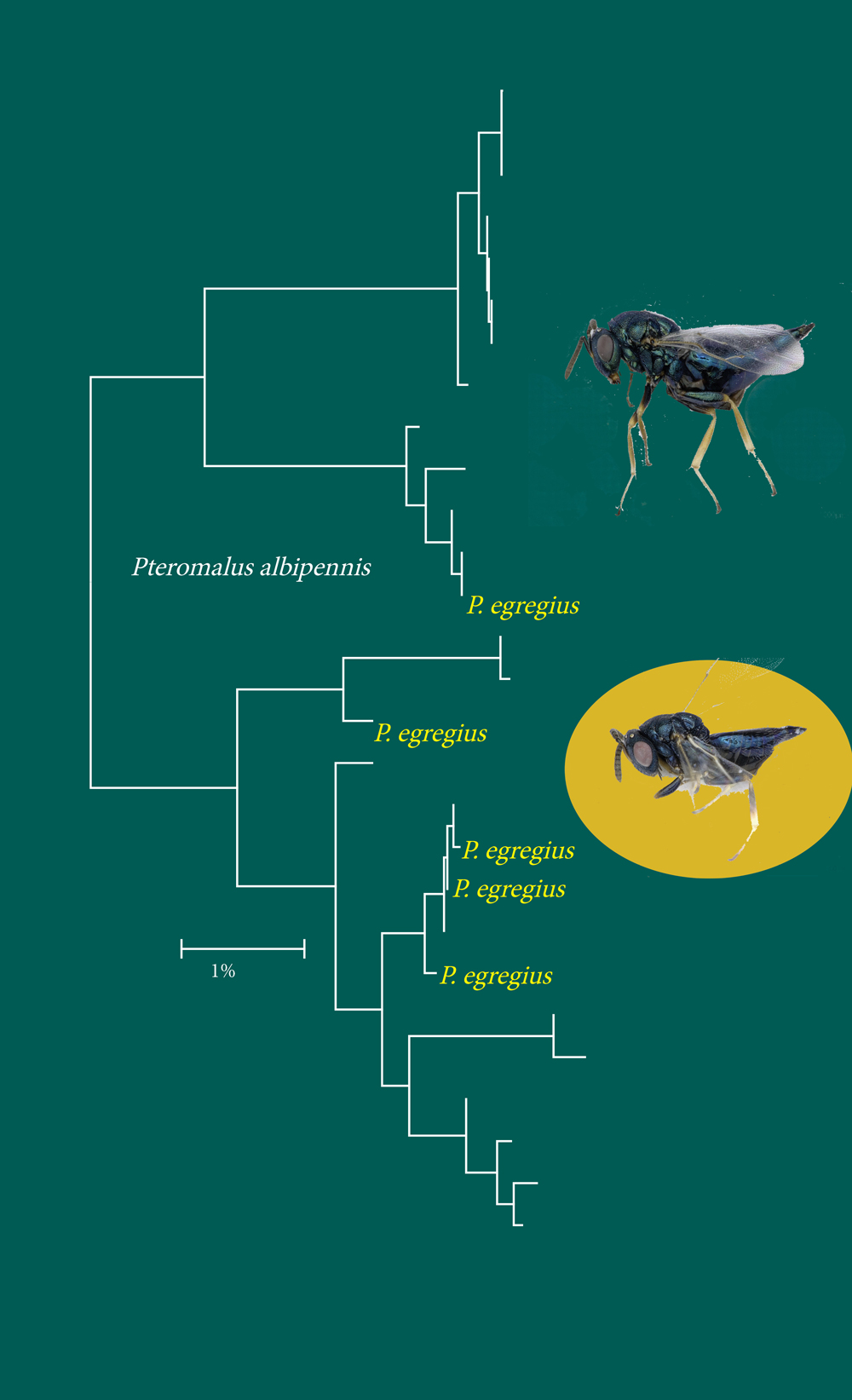
I den parasittiske vepseslekten Pteromalus (Hymenoptera) fant vi som i de andre gruppene kryptiske arter, men også det motsatte mønsteret på uoverensstemmelse: flere morfologisk beskrevne arter innenfor én strekkode-basert gruppe: P. egregius opptrer flere steder inne i P. albipennis-kladen. ITS2 ble også sekvensert, og viste det samme mønsteret. Det var stor genetisk variasjon innen P. albipennis, men variasjonen korresponderte ikke med morfologisk variasjon.
Vi kan konkludere med at DNA-strekkoding fungerer generelt godt som artsidentifisering for lav, sopp og insekter, men at det også i noen tilfiller peker på taksonomiske problemer og interessante evolusjonære mønstre.
Gunnhild Marthinsen, Mika Bendiksby, Tor Erik Brandrud, Bálint Dima, Lars Ove Hansen, Arild Johnsen, Jon Peder H. Lindemann, Einar Timdal
Disse resultatene ble presentert på 7th International Barcode of Life Conference, Sør-Afrika, 2017.

 English
English  Norwegian Bokmål
Norwegian Bokmål 








